KAPA HyperPlus试剂盒极大简化了工作流程,文库片段化和文库制备全部可在单管(Single tube)中操作完成。KAPA HyperPlus试剂盒融合了KAPA HyperPrep试剂盒的行业领先的文库构建效率和非凡的文库质量,添加了酶切片段化(enzymatic fragmentation)模块,使得工作流程更加方便、快捷。
产品特色
- 2.5 h 完成DNA片段化和文库制备
- 从1ng~1μg的灵活DNA起始量
- 扩增偏差低,测序覆盖度更统一
- 低起始量也可适用于无PCR扩增(PCR-free)的工作流程
- 针对FFPE样本,具有行业领先的文库转化率(converstion rate)和文库复杂度
- 对于DNA片段大小、接头设计和文库扩增,有多种选择方案
- 具有经过验证的自动化方案,轻松对接多种自动化平台
- 搭配KAPA接头和KAPA Pure Beads,实现文库制备全套解决方案
HyperPlus提供片段化 + 文库制备的解决方案
KAPA HyperPlus试剂盒包含低偏向性的酶切片段化DNA的模块,可以取代设备昂贵、难以自动化的机械打断DNA的方法。
- 通过更少的酶促反应及纯化步骤实现更短的实验流程及更少的手动操作步骤• KAPA RNA HyperPrep 可以作为一个单独的工作流程使用,也可以跟mRNA捕获或者
- 从起始RNA到可用于上机测序的链特异性文库只需4小时即可实现 核糖体去除(HRM)模块联合使用
- 实现整个工作流程(包括mRNA捕获或核糖体去除),可以在一个工作日内完成
- 可在自动化平台上实现高通量并稳定可靠

DNA片段大小可调整,片段化结果可重复性高
- 通过调整片段化酶切时间,文库插入片段大小可控制在150-800 bp之间
- 不同GC含量的DNA和不同的DNA起始量,都可以得到一致大小的插入片段
行业领先的文库得率
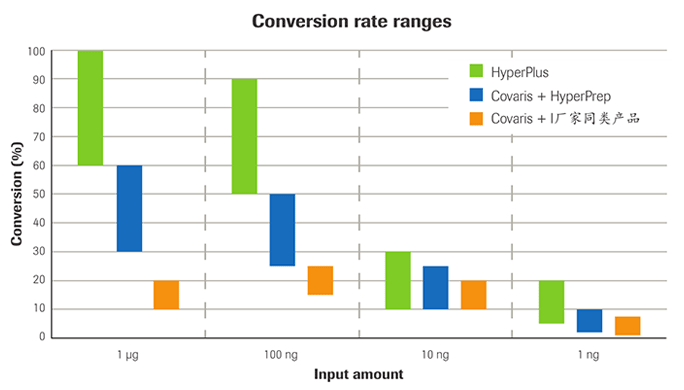
文库转化率(conversion rate)是指起始DNA分子转化为连接了接头,可以被测序的文库分子(library molecule)的百分比(%)。它是文库构建的重要评价指标,将最终决定文库的构建质量和多样性。
- DNA起始量≥100 ng时,文库转化率均可达到>80%
- 面对一系列不同质量的DNA输入都有极佳的表现,低起始量样本不必采取特殊工作流程或特殊试剂
- 高文库产出率,可允许进行DNA起始量低至50 ng的无PCR扩增(PCR-free)文库制备流程
保证优越的测序结果
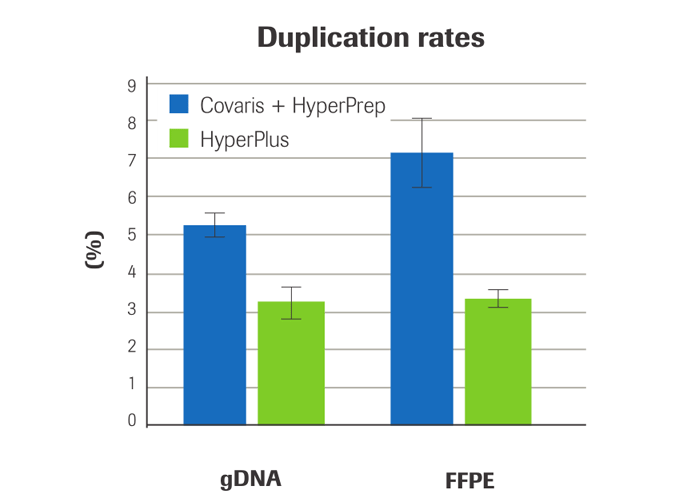
- 高转化率,使得扩增循环数减少,从而减少由扩增带来的重复率(duplication rate)
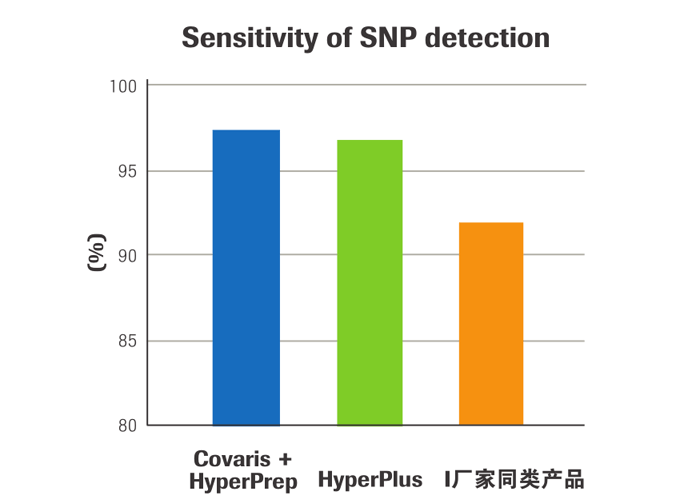
- 更高的文库多样性,更均一的测序覆盖度,使得低频突变(low-freqency mutation)的检出可信度更高
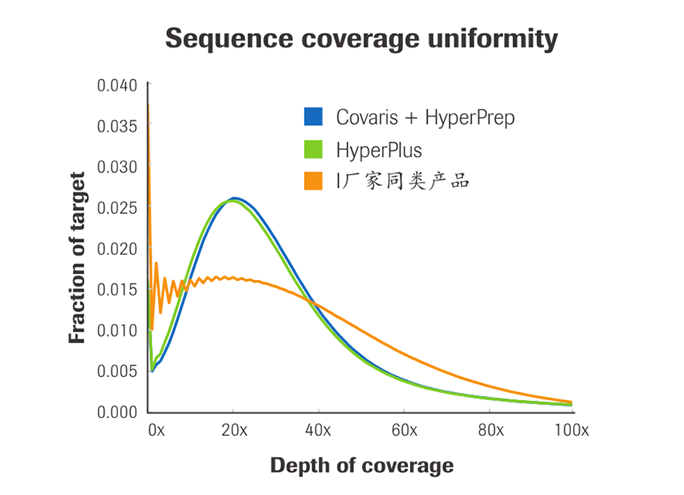
序列覆盖偏好性低
- 与tagmentation和其他酶切片段化DNA的方法相比,HyperPlus酶切模块的序列偏好性更低
- 与机械方法打断DNA的性能相当
- 由于其非常低的序列偏好性,使得测序覆盖度更均一,进而降低测序成本
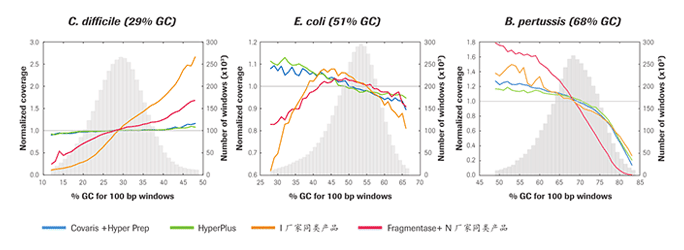
GC偏好性比较
KAPA HyperPlus文库构建试剂盒在微生物全基因组文库构建中可获得卓越的覆盖均一性及更低的GC偏好。针对3种GC含量不同的细菌,采用1nggDNA进行文库构建,文库构建试剂:KAPA HyperPrep Kit+机械打断、KAPA HyperPlus kit、N厂家dsDNA Fragmentase、N厂家Ultra DNA Library Prep kitfor illumina、I厂家XT DNA library prep kit(Illumina)。测序: Illumina MiSeq,2*300bp。GC偏差比较图(Picard软件进行分析)。灰色柱状图描述基因组GC分布,每100bp基因组进行GC含量计算。GC偏差通过在每100bp基因组上的均一化覆盖度来进行评估。如果所有的反应步骤(片段化、接头连接、文库扩增、簇扩增、测序、数据分析)均无偏差,那么均一化覆盖度应该是一条直线,且等于1。在不同GC%的物种中,可以看到KAPA HyperPlus、KAPA HyperPrep+机械打断结果较好,远远优于竞品试剂盒结果。